Código
import pandas as pd
import numpy as np
import scipy.stats as stats
from scipy.stats import expon
from scipy.stats import t
from scipy.stats import f
import plotly.express as px
import seaborn as sns
import matplotlib.pyplot as pltAllana
5 de julho de 2023
Entenda mais sobre distribuições de probabilidade com as linguagens R e Python.
Bibliotecas necessárias:
Uma distribuição de probabilidade descreve como os valores de uma variável aleatória são distribuídos. Por exemplo, sabe-se que a coleção de todos os resultados possíveis de uma sequência de lançamento de moedas seguem a distribuição binomial. Já as médias de amostras suficientemente grandes de resultados de lançamentos de dados se assemelham à distribuição normal. Como as características dessas distribuições teóricas são bem compreendidas, elas podem ser usadas para fazer inferências estatísticas em toda a população de dados.
A seguir, destrincharemos algumas das distribuições mais usadas, e como obter probabilidades usando Python e R.
Usada em casos de experimentos repetidos, onde existem dois possíveis resultados: fracasso ou sucesso. Assim, em n tentativas independentes de um experimento, cada uma tem probabilidade de sucesso p e probabilidade de fracasso 1-p.
A probabilidade de alcançar r fracassos em n elementos da amostra:
\[ \frac{n !}{(n-r) ! * r !} \]
Assim, a função de massa de probabilidade (pmf) , que é a probabilidade total de alcançar r fracassos em n elementos da amostra:
\[ \frac{n !}{(n-r) ! * r !} * p^r *(1-p)^{n-r} \]
Exemplo: Para calcular a probabilidade de obter 8 caras, a partir de um experimento com 20 tentativas, em que a probabilidade de se obter uma cara em qualquer tentativa é 0.5:
No Python, podemos usar a função “binom.pmf”, onde pmf quer dizer “probability mass function”, da biblioteca SciPy para obter a probabilidade desejada. Os argumentos passados para a função primeiro número de sucessos (8), número de tentativas (20) e por fim a probabilidade de sucesso (0.5).
Conforme abaixo:
Criamos um dataframe pandas com as probabilidades de sucesso, de 1 a 20. Após, usamos este dataframe para plotar a distribuição de probabilidade deste caso.
n = 20
p = 0.5
lista_probs=[]
n_sucessos=[]
for i in range(1,n+1):
prob=stats.binom.pmf(i, 20, 0.5)
lista_probs.append(prob)
n_sucessos.append(i)
data = {'Sucessos':n_sucessos,'Probabilidade':lista_probs}
df = pd.DataFrame(data)
fig = px.line(df, x='Sucessos', y='Probabilidade', markers=True,template='ggplot2',title='<b> Distribuição Binomial: n=20, p=0.5')
fig.show()No R, a função usada para obter a probabilidade desejada é a “dbinom”. Os argumentos no caso do exemplo serão: x=4 | size=20 | prob=0.5
Conforme abaixo:
A seguir, criamos uma tabela com as probabilidades de sucesso, de 1 a 20. Após, usamos esta tabela para plotar a distribuição de probabilidade deste caso.
n = 20
p = 0.5
binom_prob <- tibble(n_success = 1:n) %>%
mutate(prob = dbinom(n_success, size=n, prob=p))
binom_prob %>%
ggplot(aes(x=n_success, y=prob))+
geom_line()+
geom_point(size=2)+
scale_x_continuous(breaks=1:n)+
scale_y_continuous(breaks = scales::pretty_breaks(n = 5))+
labs(x= "Número de sucessos",
y= "Probabilidade",
title=paste0("Distribuição Binomial: n=20, p=0.5"))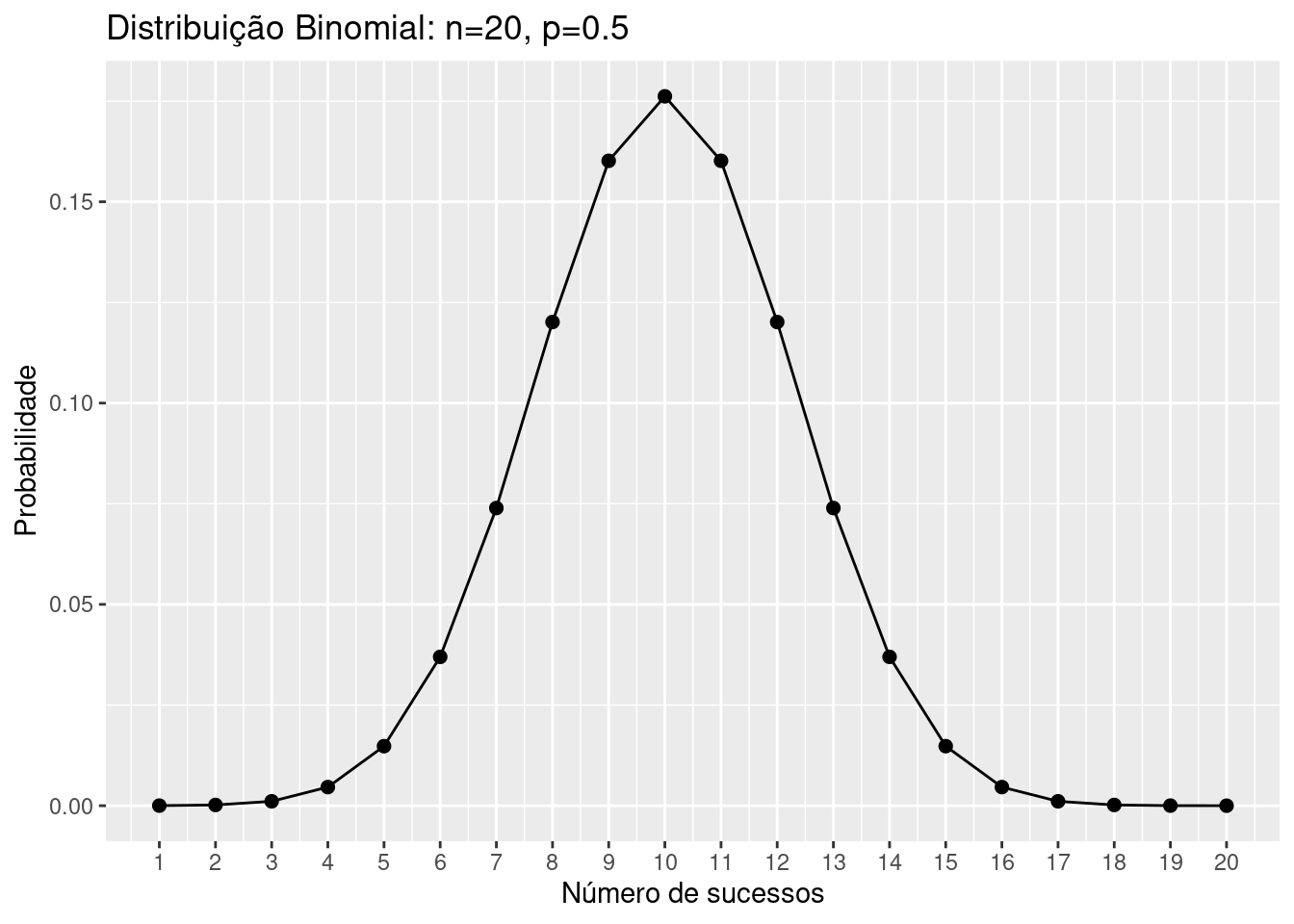
Cada distribuição de probabilidade em R está associada a quatro funções que seguem uma convenção de nomenclatura:
A função de densidade de probabilidade sempre começa com ‘d’;
A função de distribuição cumulativa sempre começa com ‘p’;
A função quantílica sempre sendo com ‘q’;
E uma função que produz variáveis aleatórias sempre começa com ‘r’.
Cada função recebe um único argumento para avaliar a função, seguido por parâmetros específicos que definem a função de distribuição específica a ser avaliada.
A Distribuição Poisson modela a probabilidade de eventos ocorrerem em um período fixo de tempo, ou seja, aqui contamos eventos que ocorrem aleatoriamente, mas a uma taxa média definida.
Exemplo: Em uma indústria, há a ocorrência de cerca de 7 ítens defeituosos por dia. Encontre a probabilidade de que, em um dia, 10 ítens apresentem defeito:
No Python, podemos usar a função “poisson.pmf”, para obter a probabilidade desejada. O primeiro argumento que passamos para a função é o 10 (queremos saber a probabilidade de observar exatamente 10 ítens defeituosos) e a taxa de defeitos por dia, que é 7. Conforme abaixo:
Criamos um dataframe pandas com as probabilidades de ítens defeituosos, de 1 a 15. Após, usamos este dataframe para plotar a distribuição de probabilidade deste caso.
n = 15
lista_probs=[]
n_prob=[]
for i in range(1,n+1):
prob=stats.poisson.pmf(i, 7)
lista_probs.append(prob)
n_prob.append(i)
data = {'Defeitos':n_prob,'Probabilidade':lista_probs}
df = pd.DataFrame(data)
fig = px.line(df, x='Defeitos', y='Probabilidade', markers=True,template='ggplot2',title='<b> Distribuição Poisson: lambda = 7')
fig.show()No R, a função usada para obter a probabilidade desejada é a “dpois”. Os argumentos no caso do exemplo serão 10 e 7.
Conforme abaixo:
Novamente, como na saída em Python, encontramos a probabilidade de cerca de 7%.
A seguir, criamos uma tabela com as probabilidades de ítens defeituosos, de 1 a 15. Após, usamos esta tabela para plotar a distribuição de probabilidade deste caso.
n = 15
pois_prob <- tibble(n = 1:n) %>%
mutate(prob = dpois(n, 7))
pois_prob %>%
ggplot(aes(x=n, y=prob))+
geom_line()+
geom_point(size=2)+
scale_x_continuous(breaks=1:n)+
scale_y_continuous(breaks = scales::pretty_breaks(n = 5))+
labs(x= "Defeitos",
y= "Probabilidade",
title=paste0("Distribuição Poisson: lambda = 7"))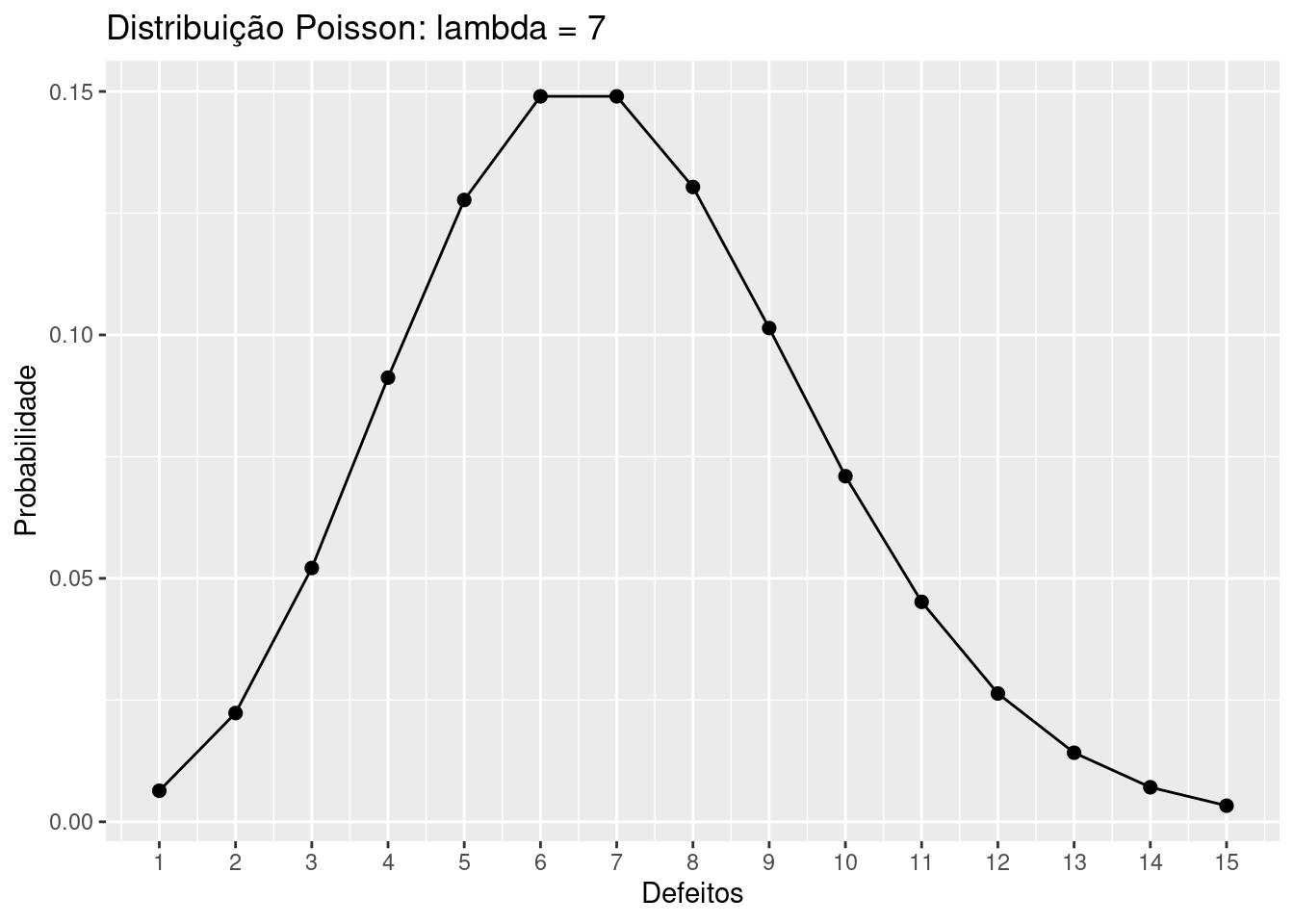
Muitas variáveis nas ciências naturais e sociais têm distribuição normal ou aproximadamente normal. Alguns exemplos são altura, peso ao nascer, satisfação no trabalho, dentre muitas outras.
Compreender as propriedades das distribuições normaisnos ajuda a usar estatísticas inferenciais para comparar diferentes grupos e fazer estimativas sobre populações usando amostras.
Características da distribuição normal:
A média, a mediana e a moda são exatamente iguais;
A distribuição é simétrica em relação à média – metade dos valores ficam abaixo da média e metade acima da média;
A distribuição pode ser descrita por dois valores: a média e o desvio padrão.
Outras características importantes:
Cerca de 68% dos valores estão dentro de 1 desvio padrão da média;
Cerca de 95% dos valores estão dentro de 2 desvios padrão da média;
Cerca de 99,7% dos valores estão dentro de 3 desvios padrão da média.
Função densidade de probabilidade:
\[ f(x)=\frac{\exp \left(-x^2 / 2\right)}{\sqrt{2 \pi}} \]
Em Python, usamos a função “norm.pdf”, para obter a distribuição desejada. Primeiro, criamos uma amostra de intervalo [-5, 5], de 0.01 em 0.01. Após, passamos para a função “norm.pdf” os parâmetros amostra, média e variância, no exemplo abaixo 0 e 1 respectivamente, conforme pode ser visto:
Podemos estar interessados em descobrir o valor de um quantil específico. Para descobrirmos o quantil 0.75, por exemplo, podemos fazer como abaixo:
Que corresponde ao valor 2.49.
[1] 0.03272988[1] 0.010934[1] 90.79184out <- seq(-9, 9, length=100)
prob <- dnorm(out)
media <- c(0, -1, 2)
dp <- c(1, 2, 3)
colors <- c("red", "blue", "darkgreen")
labels <- c("mean = 0, sd = 1", "mean = -1, sd = 2", "mean = 2, sd = 3")
colors <- c("red", "blue", "darkgreen")
plot(
out, prob, type="l", lty=2, xlab="x value",
ylab="Density", main="Comparison of Normal Distributions",
frame.plot=FALSE
)
for (i in 1:3){
lines(out, dnorm(x = out, mean = media[i], sd = dp[i]), lwd=2, col=colors[i])
}
legend("topright", inset=.05, title="Distributions",
labels, lwd=2, lty=c(1, 1, 1), col=colors)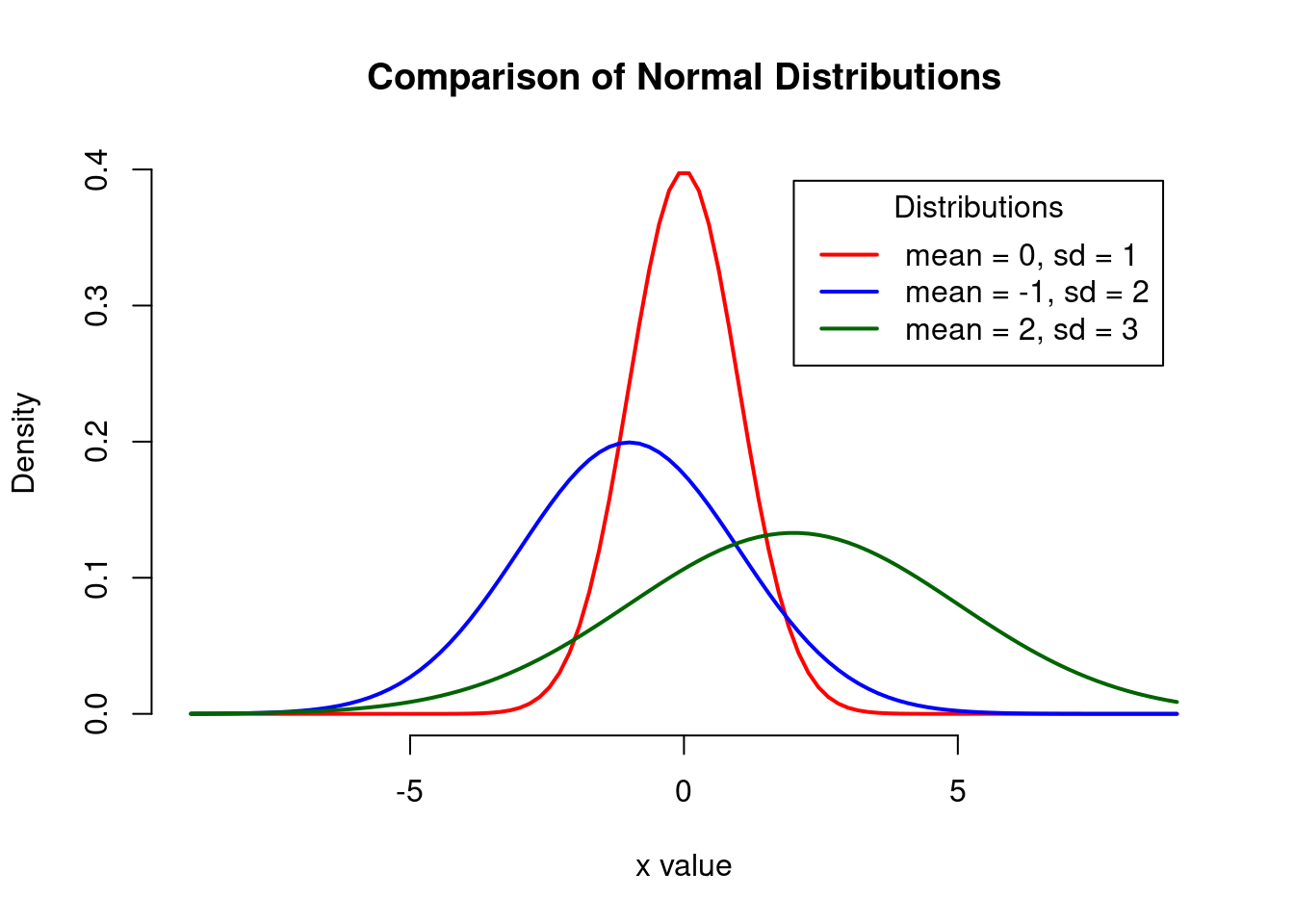
A distribuição uniforme refere-se a um tipo de distribuição de probabilidade em que todos os resultados são igualmente prováveis.
Como exemplo, poderíamos pensar na probabilidade de tirarmos de 1 a 6, ao lançarmos um dado. A probabilidade de qualquer dos resultados é de 1/6.
A distribuição exponencial é uma distribuição de probabilidade que é usada para modelar o tempo que devemos esperar até que um determinado evento ocorra.
Para gerar uma amostra exponencial, em Python, usamos a função “expon.rvs” da biblioteca SciPy. Abaixo, como exemplo, geramos uma amostra de tamanho 10, e taxa média 40.
array([ 9.02275607, 23.26908466, 8.91749082, 50.87741258,
107.57208417, 52.01864752, 11.14855552, 143.59986616,
3.62098063, 58.03659311])Para encontramos, por exemplo, a probabilidade de que x seja menor que 50 quando a taxa média for 40, podemos usar a função “expon.cdf”, conforme abaixo:
Que nos mostra uma probabilidade de cerca de 71%.
Abaixo, plotamos a distribuição de uma amostra de tamanho 250. O eixo y mostra a probabilidade daquele número de ocorrências. Mantivemos a taxa média em 40.
n = 250
lista_probs=[]
n_prob=[]
for i in range(1,n+1):
prob=expon.pdf(i, scale=40)
lista_probs.append(prob)
n_prob.append(i)
data = {'n':n_prob,'Probabilidade':lista_probs}
df = pd.DataFrame(data)
fig = px.line(df, x='n', y='Probabilidade', markers=True,template='ggplot2',title='<b> Distribuição Exponencial: lambda = 40')
fig.show()A distribuição acumulada para o exemplo ficaria como abaixo:
n = 250
lista_probs=[]
n_prob=[]
for i in range(1,n+1):
prob=expon.cdf(i, scale=40)
lista_probs.append(prob)
n_prob.append(i)
data = {'n':n_prob,'Probabilidade':lista_probs}
df = pd.DataFrame(data)
fig = px.line(df, x='n', y='Probabilidade', markers=True,template='ggplot2',title='<b> Função de Distribuição Acumulada: lambda = 40')
fig.show()[1] 0,1516327[1] 5,545177labels <- c("rate = .5", "rate = 1", "rate = 1.5")
colors <- c("red", "blue", "darkgreen")
curve(dexp(x, rate = .5), from=0, to=10, col='blue')
curve(dexp(x, rate = 1), from=0, to=10, col='red', add=TRUE)
curve(dexp(x, rate = 1.5), from=0, to=10, col='darkgreen', add=TRUE)
legend("topright", inset=.05, title="Distributions",
labels, lwd=2, lty=c(1, 1, 1), col=colors)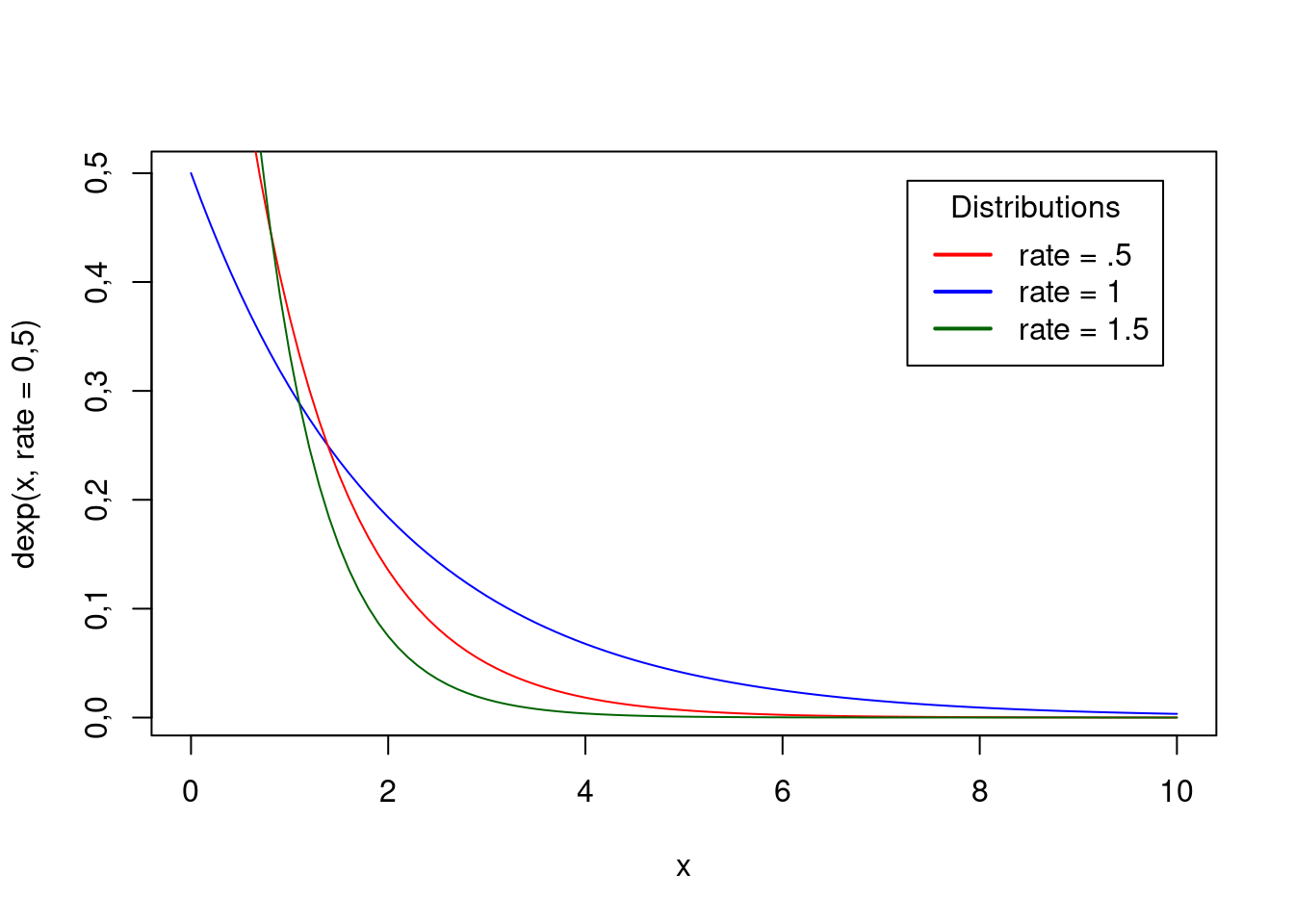
A distribuição t é uma distribuição de probabilidade semelhante à distribuição normal, porém com caudas mais “grossas”. Isso porque mais valores nesta distribuição estão localizados nas extremidades das caudas do que no centro, quando a comparamos com a distribuição normal.
Sobre os testes t, eles são usados quando é necessário comparar médias. Abaixo veremos exemplos sobre como obter p-valores, a partir de um valor hipotético da estatística de teste.
Para gerarmos uma amostra da distribuição t, podemos usar a função “t.rvs”. Os parâmetros que ela recebe são os graus de liberdade e tamanho da amostra (6 e 10, respectivamente, no exemplo).
array([-0.46425243, -0.43404586, -1.52011751, -2.07678607, -0.11229579,
-0.11445089, -0.50848308, -0.53675386, -0.27816205, 0.61669699])Suponha que realizamos um teste de hipótese unilateral e acabemos com uma estatística de teste de -1,5 e graus de liberdade = 10.
Podemos usar a seguinte sintaxe para calcular o p-valor que corresponde a esta estatística de teste:
Suponha que realizamos um teste de hipótese bicaudal e acabamos com uma estatística de teste de 2,14 e 20 graus de liberdade.
Podemos usar a seguinte sintaxe para calcular o p-valor que corresponde a esta estatística de teste:
Abaixo, com o auxílio da biblioteca do matplotlib, plotamos a distribuição normal e T, com um grau de liberdade:
# Distribuição Normal
x = np.linspace(-4, 4, 500)
y = stats.norm.pdf(x)
# Distribuição T
df = 1
y_t = stats.t.pdf(x, df)
plt.ylabel('Densidade de Probabilidade')
plt.xlabel('Desvio Padrão')
plt.plot(x, y, color='blue', label='Normal')
plt.plot(x, y_t, color='green', label=f'T, df={df}')
plt.legend()
sns.set_context('notebook')
sns.despine()
plt.show()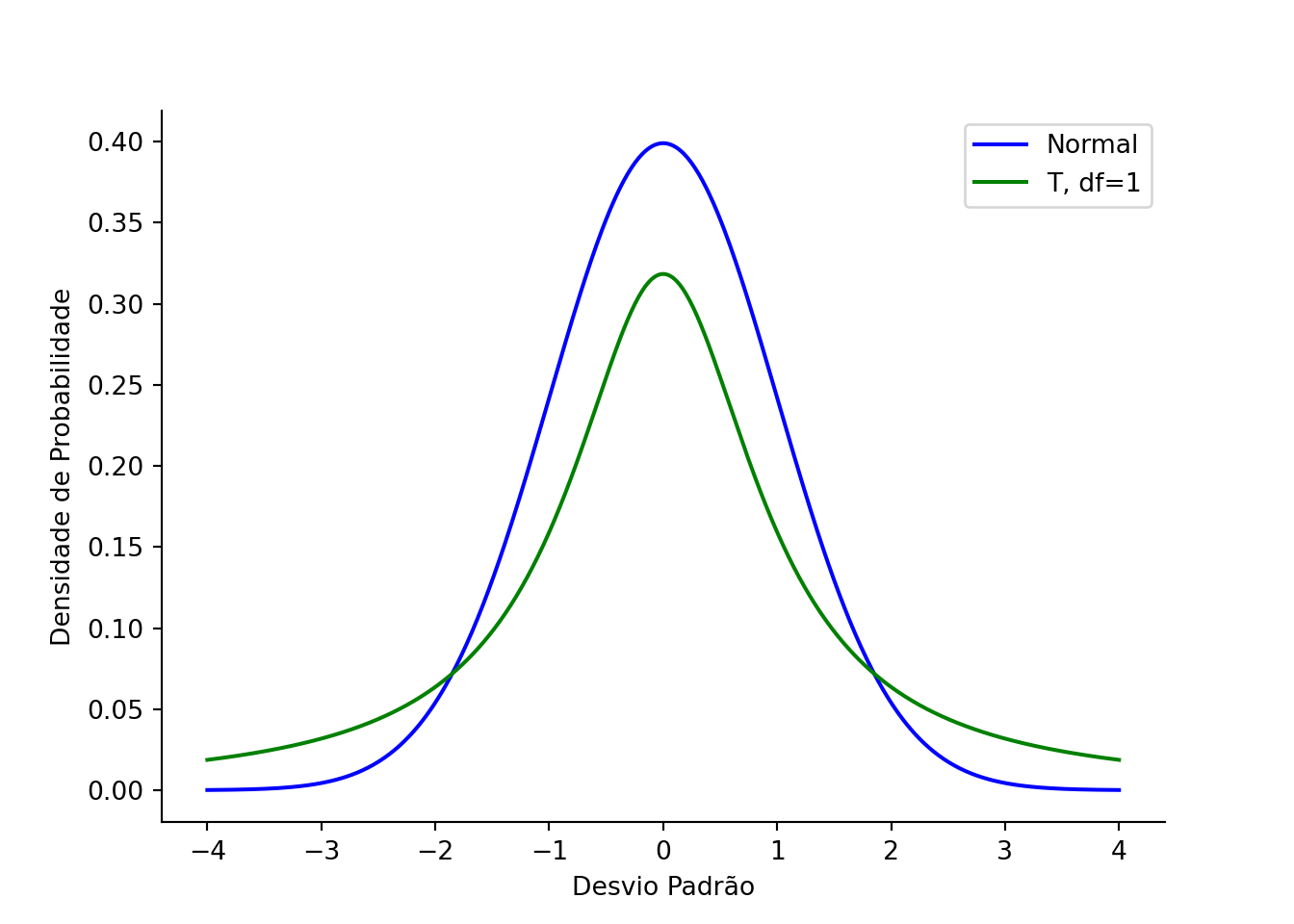
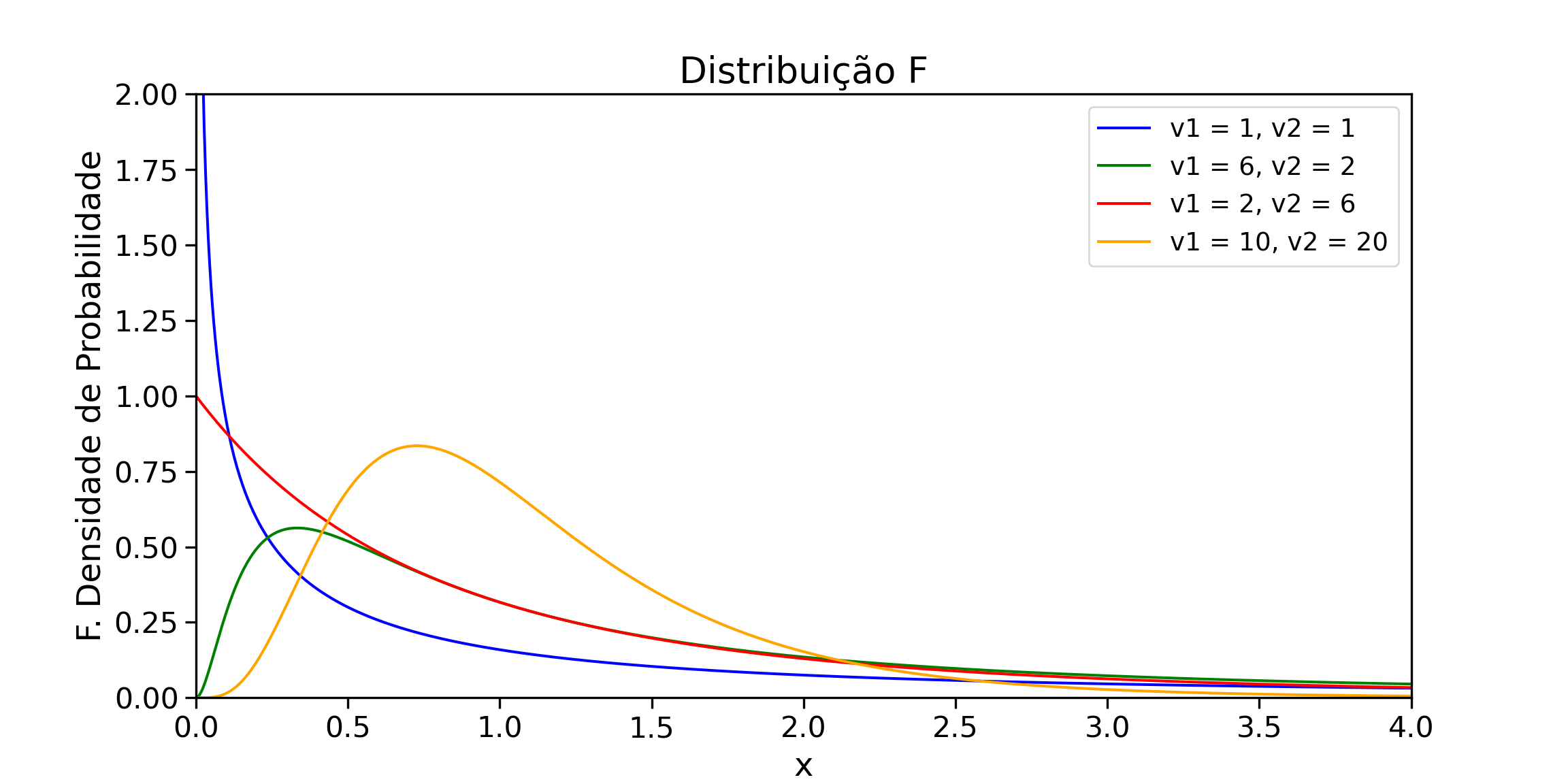
A distribuição F foi desenvolvida por Fisher para estudar o comportamento de duas variâncias de amostras aleatórias, retiradas de duas populações normais independentes. Em problemas aplicados, podemos estar interessados em saber se as variâncias populacionais são iguais ou não, com base na resposta das amostras aleatórias.
À medida que os graus de liberdade aumentam , a distribuição começa a convergir para uma distribuição normal;
A distribuição geralmente é assimétrica à direita com caudas pesadas;
É sempre positivo e contínuo.
Estas afirmações estão evidenciadas abaixo:
x = np.linspace(0, 4.5, 1000)
# Distribuição F
f1 = f(1, 1, 0)
f2 = f(6, 2, 0)
f3 = f(2, 6, 0)
f4 = f(10, 20, 0)
# plot Distribuição F
plt.figure(figsize=(12, 6))
plt.plot(x, f1.pdf(x), label = 'v1 = 1, v2 = 1', color = 'blue')
plt.plot(x, f2.pdf(x), label = 'v1 = 6, v2 = 2', color = 'green')
plt.plot(x, f3.pdf(x), label = 'v1 = 2, v2 = 6', color = 'red')
plt.plot(x, f4.pdf(x), label = 'v1 = 10, v2 = 20', color = 'orange')
plt.xlim(0, 4)(0.0, 4.0)(0.0, 2.0)(array([0. , 0.5, 1. , 1.5, 2. , 2.5, 3. , 3.5, 4. ]), [Text(0.0, 0, '0.0'), Text(0.5, 0, '0.5'), Text(1.0, 0, '1.0'), Text(1.5, 0, '1.5'), Text(2.0, 0, '2.0'), Text(2.5, 0, '2.5'), Text(3.0, 0, '3.0'), Text(3.5, 0, '3.5'), Text(4.0, 0, '4.0')])(array([0. , 0.25, 0.5 , 0.75, 1. , 1.25, 1.5 , 1.75, 2. ]), [Text(0, 0.0, '0.00'), Text(0, 0.25, '0.25'), Text(0, 0.5, '0.50'), Text(0, 0.75, '0.75'), Text(0, 1.0, '1.00'), Text(0, 1.25, '1.25'), Text(0, 1.5, '1.50'), Text(0, 1.75, '1.75'), Text(0, 2.0, '2.00')])